2024年5月14日,上海交通大学生命科学技术学院余祥长聘教轨副教授课题组和辰山植物园杨俊/王红霞团队合作在国际知名期刊《Nature Communications》发表题为“Transfer learning enables identification of multiple types of RNA modifications using nanopore direct RNA sequencing”的研究论文。作者结合纳米孔RNA直接测序技术和深度学习方法开发了可同时检测多种类型RNA修饰的迁移学习模型TandemMod(图1)。上海交通大学生命科学技术学院博士后吴优为论文第一作者,上海交通大学生命科学技术学院余祥长聘教轨副教授、辰山植物园王红霞副研究员和杨俊研究员为该论文通讯作者。
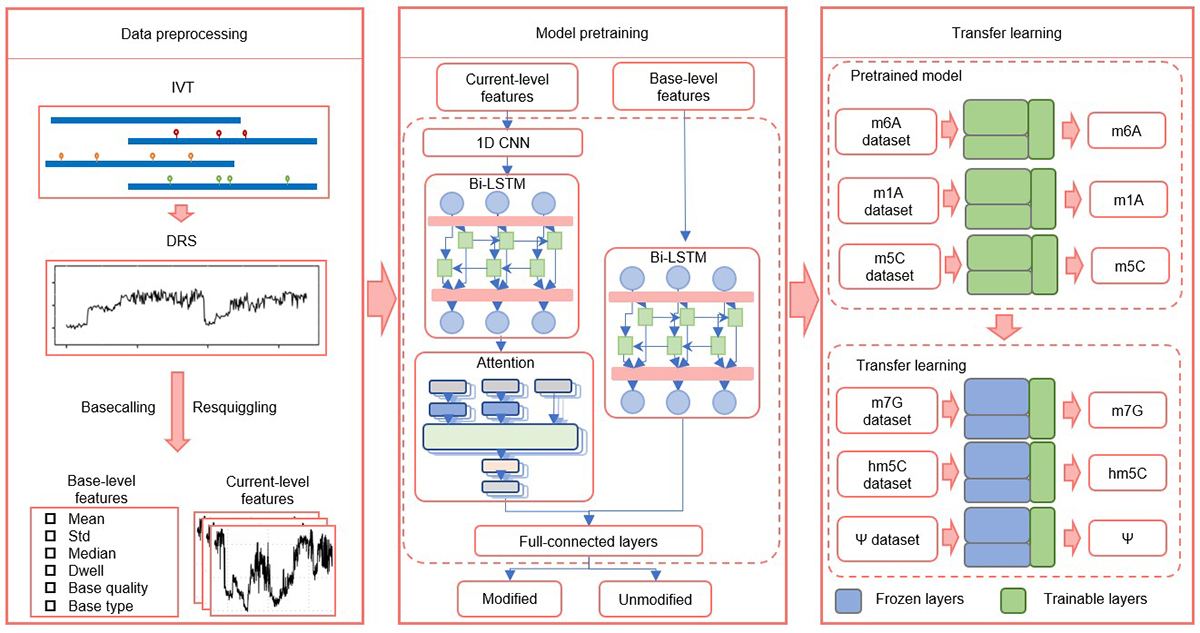
图1. 基于纳米孔RNA直接测序数据搭建TandemMod深度学习框架的示意图。
自然界RNA中存在160种以上的修饰类型。近年的研究揭示mRNA上的m6A、m5C、m1A和Ψ等修饰具有重要的生物学功能。结合抗体捕获、化学突变和限制性酶切等方法的高通量测序技术被广泛用于对mRNA某一类型修饰位点的鉴定。然而,如何在同一条mRNA转录本中同时鉴定多种不同类型的修饰位点还存在巨大挑战。相较于二代测序技术,纳米孔RNA直接测序 (Direct RNA Sequencing, DRS)能够对带有polyA尾的全长mRNA进行直接测序,mRNA穿过纳米孔时产生的电信号差异可用于推断单条mRNA上每个碱基的类别及其修饰信息(图2)。因此,结合机器学习算法,理论上DRS可实现在单条mRNA水平、以单碱基分辨率检测多种不同类型的碱基修饰。但是,目前纳米孔的电信号特征与天然mRNA上的哪些修饰碱基相对应还缺乏准确的标签,这制约了RNA修饰检测模型的训练和建立。
图2. RNA通过纳米孔时产生电信号信息的示意图。
该研究中作者创新性地利用带有T7启动子的水稻cDNA文库体外转录出了具备不同修饰标签的数千种mRNA转录本,加polyA尾后通过DRS创建了分别带有未修饰碱基、m6A修饰、m5C修饰和m1A修饰等精准标签的体外表观转录组 (IVET)数据集(图3)。在此基础上,作者以每5个碱基分配到的电信号及其统计特征作为输入,训练并搭建了一个具备1维卷积神经网络 (1D CNN)、双向长短时记忆(Bi-LSTM)和注意力机制(Attention)的TandemMod深度学习框架(图1)。
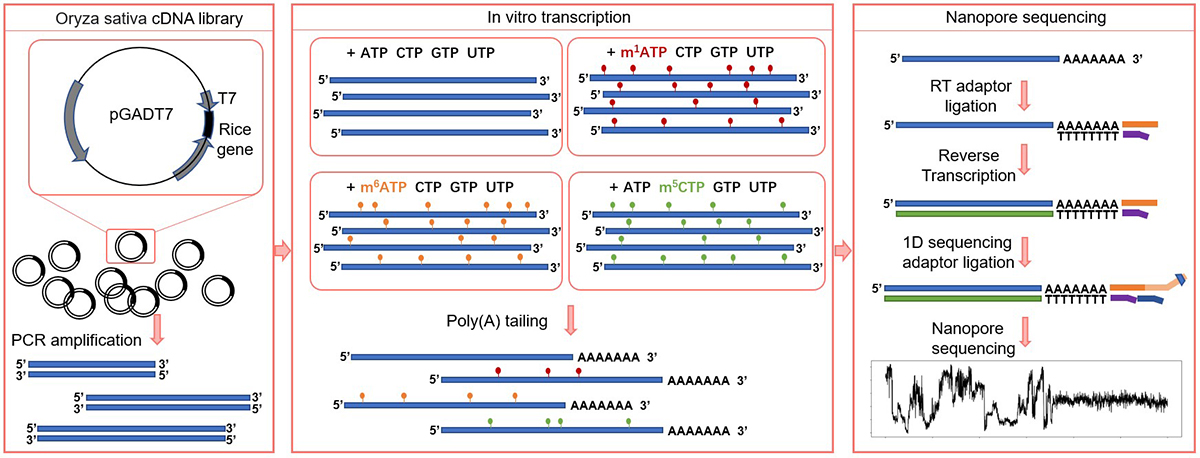
图3. 利用水稻cDNA文库体外转录带有m6A、m1A和m5C标签的转录本、并通过DRS创建体外表观转录组的示意图。
作者首先将TandemMod模型与目前基于DRS预测m6A和m5C的主流算法进行比较,验证了TandemMod模型在体外转录本及细胞体内转录本DRS数据中预测RNA修饰位点的优越性能(图4)。在正常人类细胞中检测到排名前五的基序中有4个是已知的DRACH,在m6A甲基化酶敲除的细胞中m6A修饰率显著下降。
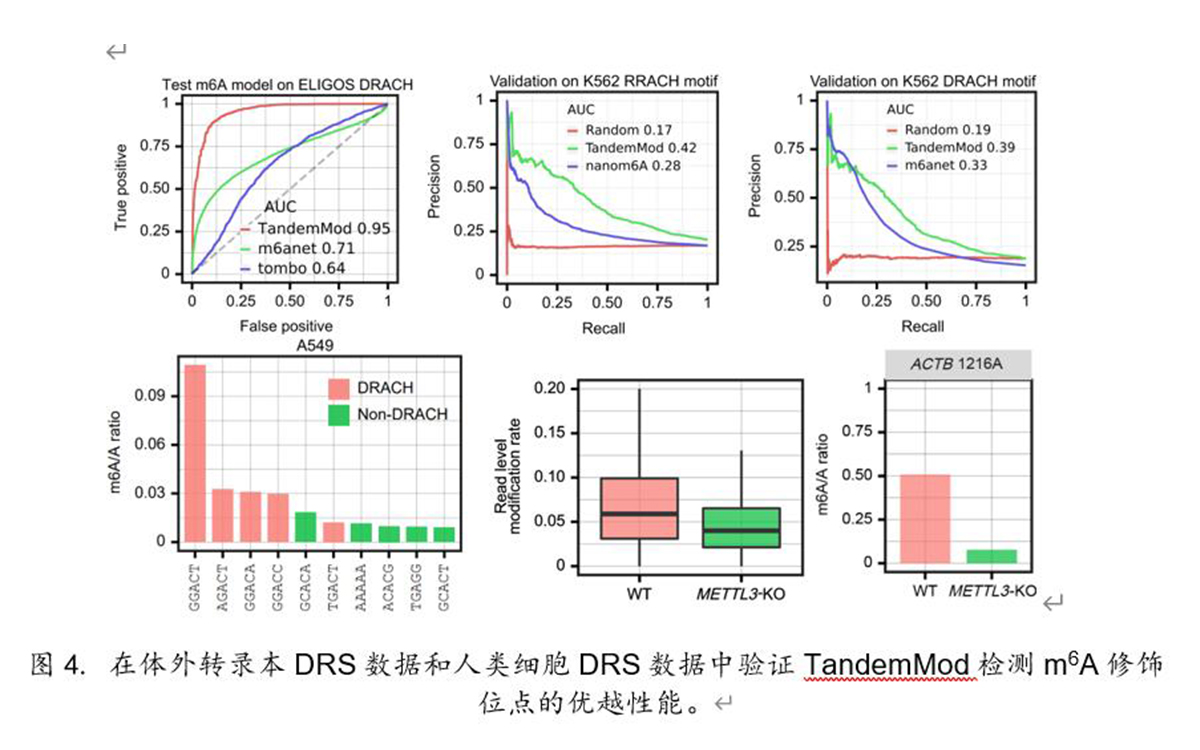
图4. 在体外转录本DRS数据和人类细胞DRS数据中验证TandemMod检测m6A修饰位点的优越性能。
进一步,作者通过迁移学习将TandemMod用于检测mRNA中的m7G、 hm5C、 Ψ 和Inosine等多种类型的RNA修饰位点,揭示了迁移学习可在保证同等性能的条件下显著减少训练集数据量和模型训练时间等计算成本(图5)。最后,作者通过TandemMod绘制了水稻幼苗在高盐胁迫下m6A、m5C和Ψ的表观修饰图谱,并揭示了mRNA中m6A和m5C的共修饰及其在高盐环境下的修饰率变化(图6)。
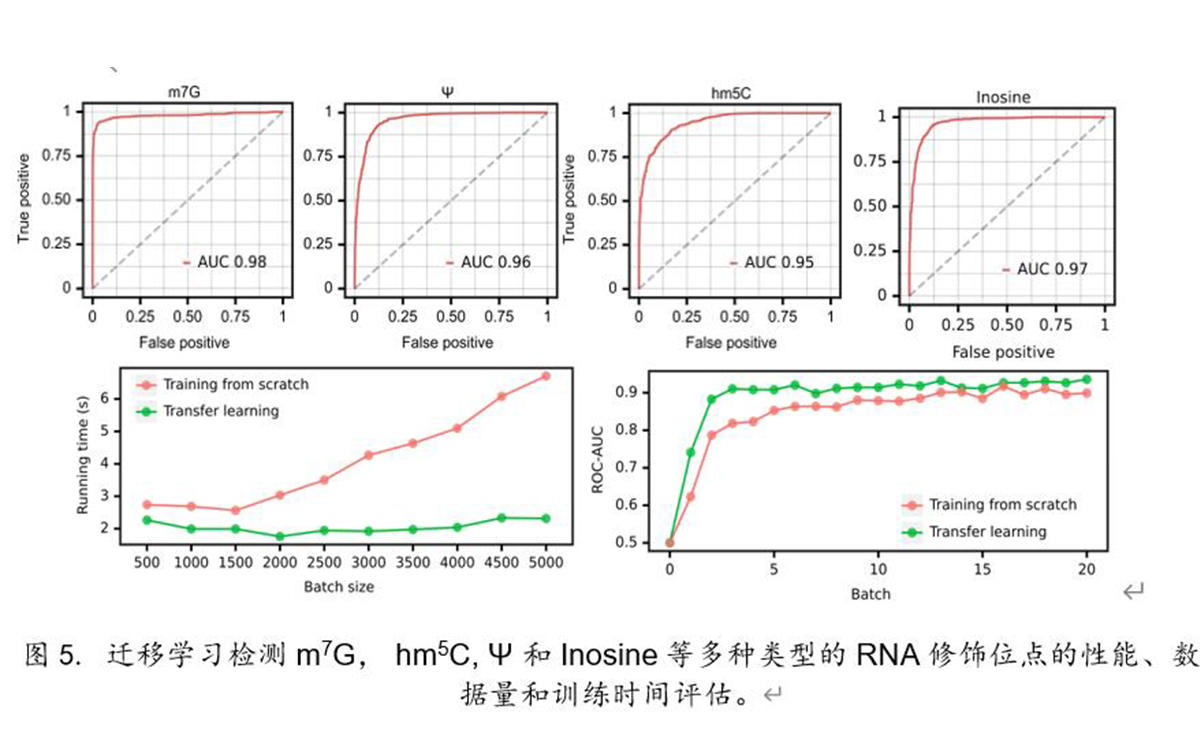
图5. 迁移学习检测m7G, hm5C, Ψ 和Inosine等多种类型的RNA修饰位点的性能、数据量和训练时间评估。
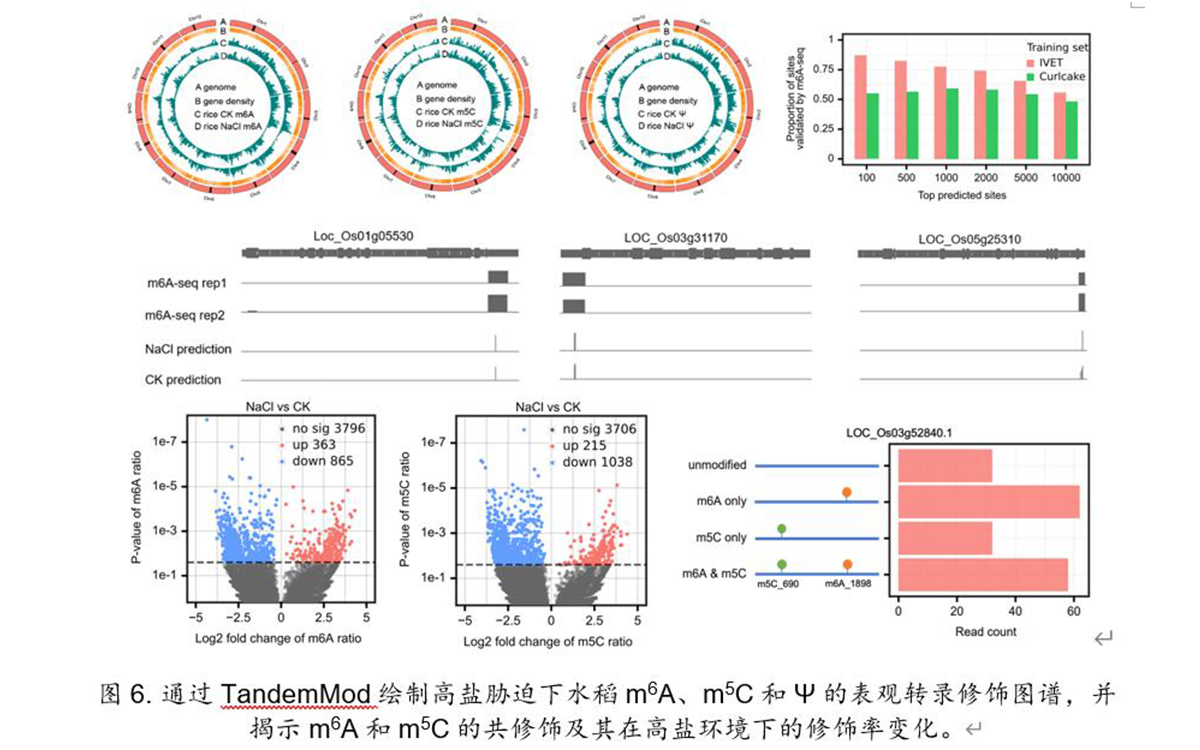
图6. 通过TandemMod绘制高盐胁迫下水稻m6A、m5C和Ψ的表观转录修饰图谱,并揭示m6A和m5C的共修饰及其在高盐环境下的修饰率变化。
综上,作者创建的体外表观转录组 (IVET) 将为基于纳米孔RNA单分子测序鉴定RNA修饰的机器学习方法提供基准训练数据集,开发的可迁移深度学习模型TandemMod将为动植物和微生物体内多种类型的RNA修饰位点鉴定及表观转录组研究提供重要的技术支撑。同时TandemMod还可用于检测RNA疫苗等人工修饰的RNA。
该研究得到了国家自然科学基金、上海市浦江人才和上海市科技创新行动计划的支持。感谢上海辰山植物园研究平台在纳米孔测序的帮助以及上海交通大学超算平台提供的计算资源。
论文链接:https://www.nature.com/articles/s41467-024-48437-4
文:王红霞